Cell Metab | 被忽视的衰老“元凶”:药物干预蛋白翻译保真度可延长机体寿命
相较于大量探究DNA突变对机体衰老和疾病影响的研究而言,人们对蛋白质翻译错误在其中的作用仍知之甚少。然而,蛋白质翻译错误的频率与DNA突变相比却高出几个数量级。最近,翻译保真度与衰老之间的联系在一系列针对酵母的研究中得到证实,例如线粒体核糖体的低精确突变体会导致酵母寿命缩短,相反,超精确突变体则显示延长的寿命和改善的蛋白稳态[1]。另外,啮齿动物细胞也提供了关于翻译准确性和寿命的相关性的证据[2]。但尚未研究如何调节蛋白质翻译保真度以延长多细胞生物寿命。
蛋白翻译保真度由核糖体解码中心决定,在最初的tRNA选择过程中,同源氨酰tRNA诱导小核糖体亚基中的域关闭,从而激活EF-Tu/EF1A以进行GTP水解,随后便将正确的氨酰-tRNA插入到肽基转移酶中心。在这些步骤中,导致翻译错误的主要因素是tRNA的错酰化和肽基转移到核糖体A位点错配的tRNA上[3, 4]。基于这些研究结果,研究人员推断是否可以通过提高蛋白翻译保真度来提供对多细胞生物的抗衰老干预?
2021年9月14日,来自英国UCL癌症研究所的Ivana Bjedov团队在Cell Metabolism杂志上发表了一篇题为 Increased fidelity of protein synthesis extends lifespan 的文章,在这项研究中,研究人员通过对核糖体解码中心的一种关键蛋白质RPS23的系统发育分析发现,引入其单个氨基酸的突变来提高翻译保真度足以改善酵母、线虫和果蝇的健康和寿命。此外,该研究还证明雷帕霉素、Torin1 和曲美替尼等抗衰老药物通过减少翻译错误来延长生物体寿命的统一作用模式, 为通过翻译准确性干预措施来延缓衰老进程提供了有力证据。

考虑到RPS23蛋白对翻译准确性的重要性,研究人员首先利用不同的数据库对从古生菌到真核生物的RPS23进行了无偏系统发育分析,并鉴定出在整个进化过程中显着保守的赖氨酸残基,而唯一例外是在生活于高温和酸性环境等极端条件下的超嗜热古细菌中,该赖氨酸被精氨酸所取代。为了研究这一点突变对于翻译保真度的意义,研究人员在果蝇rps23中引入了这一K60R突变,与对照相比,与年龄相关的翻译错误明显减少,但并不会影响整体的翻译水平。
有意思的是,这一点突变存在于生活在极端条件下的超嗜热古细菌中,是不是意味着该突变能够赋予这种古生菌更强大的热应激耐受性?与这一猜想一致, RPS23 K60R突变显著提高了酵母、线虫和果蝇在热应激下的存活率,提示维持蛋白稳态的能力得到增强。既然这个突变能给生物体带来这种增益,为什么并没有在自然界中被优先选择呢?研究人员发现,这三种生物体均表现出生长发育迟缓表型,这也可以解释R残基仅存在于超嗜热古细菌中的现象,在极端条件下,增加的翻译保真度可能代表一种强大的选择压力。RPS23 K60R不仅赋予了这三种生物体耐热特性,同时还伴随着更强壮的体魄和延长的寿命。例如,负趋地性或攀爬试验结果显示K60R改善了果蝇的攀爬能力,携带K60R的线虫和果蝇还表现出更缓慢的生殖能力衰退。
截至目前,研究最为充分的抗衰老药物之一,mTOR抑制剂雷帕霉素已在体外哺乳动物细胞中被证实能够减少翻译错误[5]。那么其他抗衰药物是否也通过类似途径来发挥作用?为了验证这个想法,研究人员在果蝇S2R+细胞中通过双荧光素酶报告系统检测两种常见的翻译错误(终止密码子通读和氨基酸错误掺入)。可以肯定的是,雷帕霉素提高了果蝇S2R+细胞的翻译保真度,并降低了终止密码子通读和错误掺入。不仅如此,曲美替尼和Torin1两种抗衰药物也能通过类似方式提高翻译保真度,提示这些抗衰药物的统一作用机制(图1)。进一步地,若将雷帕霉素应用于这三种生物体活体,可以发现雷帕霉素可以延长野生型酵母和果蝇的寿命,而轻微延长携带RPS23 K60R突变体的生物体寿命,导致最终野生型和突变型寿命相似。而在线虫中,仅延长野生型寿命,并未进一步延长突变型寿命。这些结果提示这三种生物体寿命对于蛋白翻译保真度的依赖性。
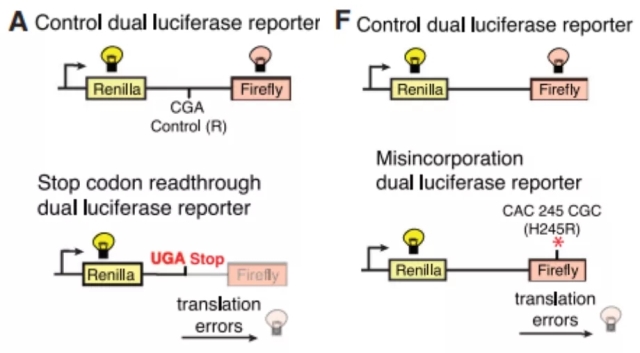
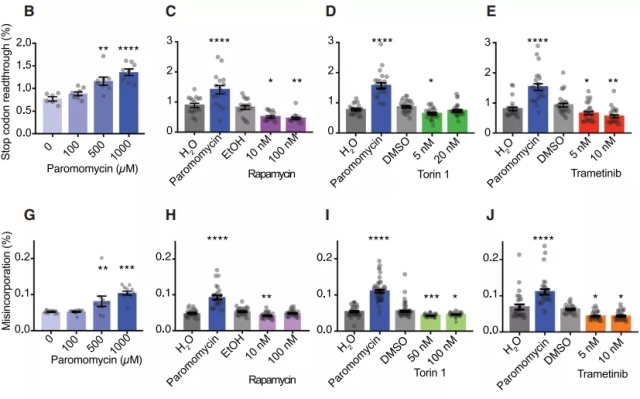
图1. 用于检测两种常见翻译错误的双荧光素酶报告系统模式图和不同抗衰药物的效用检测
此前,DNA损伤通常被认为是机体衰老的“罪魁祸首”,而这项研究为衰老机制的理解增加了另一个维度。总的来说,这项研究表明,筛选具有减少衰老过程中蛋白翻译错误潜力的化合物不失为延缓衰老,尤其对于主要受蛋白稳态恶化影响的神经退行性疾病而言的一种有效策略。
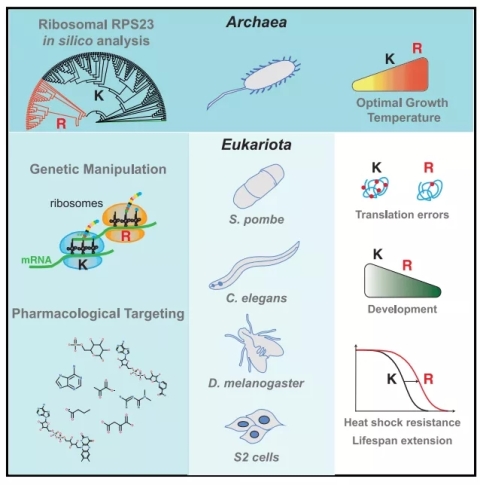
(来源:BioArt)
参考文献:
1. Suhm, T., Kaimal, J.M., Dawitz, H., Peselj, C., Masser, A.E., Hanze′ n, S., Ambrozic, M., Smialowska, A., Bjo ¨ rck, M.L., Brzezinski, P., et al. (2018). Mitochondrial translation efficiency controls cytoplasmic protein homeostasis. Cell Metab. 27, 1309–1322.e6.
2. Suhm, T., Kaimal, J.M., Dawitz, H., Peselj, C., Masser, A.E., Hanze′ n, S., Ambrozic, M., Smialowska, A., Bjo ¨ rck, M.L., Brzezinski, P., et al. (2018). Mitochondrial translation efficiency controls cytoplasmic protein homeostasis. Cell Metab. 27, 1309–1322.e6.
3. Ogle, J.M., and Ramakrishnan, V. (2005). Structural insights into translational fidelity. Annu. Rev. Biochem. 74, 129–177.
4. Zaher, H.S., and Green, R. (2009). Fidelity at the molecular level: lessons from protein synthesis. Cell 136, 746–762.
5. Johnson, S.C., Rabinovitch, P.S., and Kaeberlein, M. (2013). mTOR is a key modulator of ageing and age-related disease. Nature 493, 338–345.
链接:https://mp.weixin.qq.com/s/GM5lIDfIOjLvnoKkIMga2g















